class: center, middle, inverse, title-slide # Analyse de la covariance ### Marie-Pierre Etienne ### <a href="mailto:marie-pierre.etienne@agrocampus-ouest.fr" class="email">marie-pierre.etienne@agrocampus-ouest.fr</a> ### 2021/11/19 (updated: 2023-09-08) --- --- name: ancova # Comparaison de droite de régression : modèle d'analyse de la covariance -- ## Cadre général C'est la situation où * la variable à expliquer `\(Y\)` est quantitative * Les variables explicatives sont qualitatives et quantitatives ``` ## `geom_smooth()` using formula 'y ~ x' ``` ``` ## Warning: Removed 2 rows containing non-finite values (stat_smooth). ``` ``` ## Warning: Removed 2 rows containing missing values (geom_point). ``` 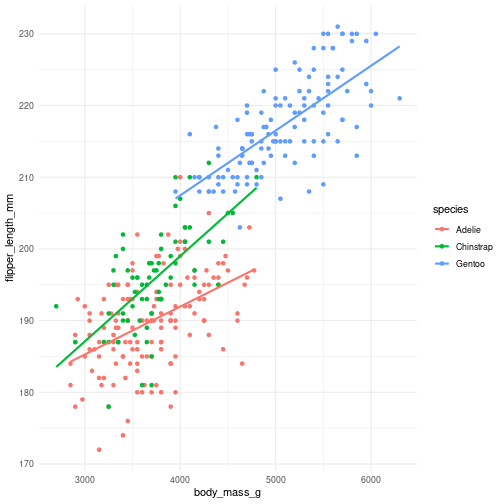<!-- --> --- template: ancova ## Le modèle `$$M_{comp} : \class{alea}{Y_{ik}} = \class{rouge}{\mu} + \class{rouge}{\alpha_i} + \class{rouge}{\beta}x_{ik} +\class{rouge}{\gamma_i} x_{ik} + \class{alea}{E_{ik}},\quad \class{alea}{E_{ik}}\overset{ind}{\sim}\mathcal{N}(0, \class{rouge}{\sigma^2}),$$` avec - `\(i=1,\ldots,I\)` le niveau du facteur 1, - `\(x_{ik}\)` la mesure de la variable quantitative `\(x\)` sur l'individu `\(k\)` du groupe `\(i\)`, - `\(\class{rouge}{\mu}\)` l'ordonnée à l'origine de référence, - `\(\class{rouge}{\alpha_i}\)` l'effet différentiel de l'espèce `\(i\)` sur l'ordonnée à l'origine, ( `\(\alpha_1=0\)` ) - `\(\class{rouge}{\beta}\)` l'effet de la variable `\(x\)` pour le groupe de référence, - `\(\class{rouge}{\gamma_i}\)` l'effet différentiel de la variable `\(x\)` pour le groupe `\(i\)` par rapport au groupe de référence ( `\(\gamma_1=0\)` ), - `\(\class{rouge}{\sigma^2}\)` la variance commune à tous les groupes. ### Nombre de paramètres du modèle - `\(2I\)` paramètres de moyenne `\((\class{rouge}{(\mu, \alpha_2, \ldots, \alpha_I, \beta, \gamma_2, \ldots, \gamma_I)}\)`; - 1 paramètre de variance `\(\class{rouge}{\sigma^2}\)` --- name: test_complet # Tests dans le modèle d'analyse de la covariance -- ## Test du modèle complet Pour répondre à la question "La longueur des ailes des manchots est elle liée à leur masse et/ou à leur espèce ?" `$$\begin{align} H_0 & =\left \lbrace \mbox{La longueur des ailes est la même en moyenne quelque soit la masse ou l'espèce du manchot }\right\rbrace\\ & =\left \lbrace \mbox{pour tout }i, \alpha_i =0, \gamma_i = 0 \mbox{ et }\beta=0 \right\rbrace\\ & =\left \lbrace M_{comp} \mbox{ est équivalent à } M0 \right\rbrace. \end{align}$$` `\(H_1\)` prend les formes équivalentes suivantes `$$\begin{align} H_1 & =\left \lbrace \mbox{la longueur des ailes varie selon la masse ou l'espèce }\right\rbrace\\ & =\left \lbrace \mbox{Il existe un }i, \alpha_i \ne 0 \mbox{ ou un } \gamma_i \ne 0 \mbox{ ou } \beta \ne 0 \right\rbrace\\ & =\left \lbrace M_{comp} \mbox{ est préférable à } M0 \right\rbrace. \end{align}$$` --- template: test_complet ## Test du modèle complet ### Statistique de test $$ F = \frac{\frac{RSS_0-RSS}{2I-1}}{\frac{RSS}{n-2I}} \underset{H_0}{\sim} \mathcal{F}(2I-1, n-2I) $$ --- template: test_complet ## Test du modèle complet sur l'exemple manchot ```r Mcomp <- lm(flipper_length_mm ~ species + body_mass_g + species:body_mass_g, data =penguins) M0 <- lm(flipper_length_mm ~ 1, data =penguins) anova(M0, Mcomp) ``` ``` ## Analysis of Variance Table ## ## Model 1: flipper_length_mm ~ 1 ## Model 2: flipper_length_mm ~ species + body_mass_g + species:body_mass_g ## Res.Df RSS Df Sum of Sq F Pr(>F) ## 1 341 67427 ## 2 336 9611 5 57815 404.24 < 2.2e-16 *** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ``` --- template: test_complet ## Test de l'égalité des pentes -- `$$\begin{align} H_0 & =\left \lbrace \mbox{La longueur des ailes augmente de la même manière avec le poids quelque soit l'espèce }\right\rbrace\\ & =\left \lbrace \mbox{pour tout }i, \gamma_i = 0 \right\rbrace\\ & =\left \lbrace M_{comp} \mbox{ est équivalent à } M_{12} \right\rbrace. \end{align}$$` `$$M_{12} : \class{alea}{Y_{ik}} = \class{rouge}{\mu} + \class{rouge}{\alpha_i} + \class{rouge}{\beta}x_{ik} + \class{alea}{E_{ik}},\quad \class{alea}{E_{ik}}\overset{ind}{\sim}\mathcal{N}(0, \class{rouge}{\sigma^2})$$` -- `$$\begin{align} H_1 & =\left \lbrace \mbox{L'augmentation de longueur des ailes avec le poids n'est pas la même pour toutes les espèces}\right\rbrace\\ & =\left \lbrace \mbox{Il existe un }i,\gamma_i \ne 0 \right\rbrace\\ & =\left \lbrace M_{comp} \mbox{ est préférable à } M_{12} \right\rbrace. \end{align}$$` --- template: test_complet ## Test de l'égalité des pentes ### Statistique de test -- `$$F = \frac{\frac{RSS_{12}-RSS}{I-1}} {\frac{RSS}{n-2I}} \underset{H_0}{\sim} \mathcal{F}(I-1, n-2I)$$` -- 2 situations : * si les pentes sont différentes, on a mis en évidence un effet de l'espèce et un effet de la masse ne serait ce qu'au travers de leur interaction. * si les pentes sont identiques, le modèle `\(M_{12}\)` devient alors le modèle le plus complet considéré et on teste si la pente est différente de 0 et on teste si les ordonnées à l'origine sont identiques. Quels modèles peut on comparer ? --- template: test_complet ## Test de l'égalité des pentes sur l'exemple manchots ```r M12 <- lm(flipper_length_mm ~ species + body_mass_g, data =penguins) anova(M12, Mcomp) ``` ``` ## Analysis of Variance Table ## ## Model 1: flipper_length_mm ~ species + body_mass_g ## Model 2: flipper_length_mm ~ species + body_mass_g + species:body_mass_g ## Res.Df RSS Df Sum of Sq F Pr(>F) ## 1 338 9839.1 ## 2 336 9611.2 2 227.91 3.9837 0.0195 * ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ``` --- template: test_complet Que teste-t-on avec cette commande ? ```r library(car) ``` ``` ## Warning: package 'car' was built under R version 4.0.5 ``` ``` ## Loading required package: carData ``` ``` ## ## Attaching package: 'car' ``` ``` ## The following object is masked from 'package:dplyr': ## ## recode ``` ``` ## The following object is masked from 'package:purrr': ## ## some ``` ```r Anova(Mcomp) ``` ``` ## Anova Table (Type II tests) ## ## Response: flipper_length_mm ## Sum Sq Df F value Pr(>F) ## species 6411.2 2 112.0661 <2e-16 *** ## body_mass_g 5114.2 1 178.7885 <2e-16 *** ## species:body_mass_g 227.9 2 3.9837 0.0195 * ## Residuals 9611.2 336 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ``` --- name: ajuste # Comparer les espèces à poids équivalents La répondre à la question "Les longueurs des ailes des manchots des différentes espèces sont-elles comparables" Pour rendre la comparaison juste, il faut choisir un poids de référence. -- Moyenne ajustée `$$\hat{\mu}_i = \mu + \alpha_i + (\beta+\gamma_i) \bar{x}$$` -- ```r library(emmeans) ``` ``` ## Warning: package 'emmeans' was built under R version 4.0.5 ``` ``` ## ## Attaching package: 'emmeans' ``` ``` ## The following object is masked from 'package:devtools': ## ## test ``` ```r mean_length <- emmeans(object = Mcomp, specs = ~ species) ``` ``` ## NOTE: Results may be misleading due to involvement in interactions ``` ```r pairs(mean_length, adjust = "bonf") ``` ``` ## contrast estimate SE df t.ratio p.value ## Adelie - Chinstrap -8.10 1.21 336 -6.678 <.0001 ## Adelie - Gentoo -15.99 1.16 336 -13.733 <.0001 ## Chinstrap - Gentoo -7.88 1.41 336 -5.582 <.0001 ## ## P value adjustment: bonferroni method for 3 tests ``` ```r plot(mean_length) ``` 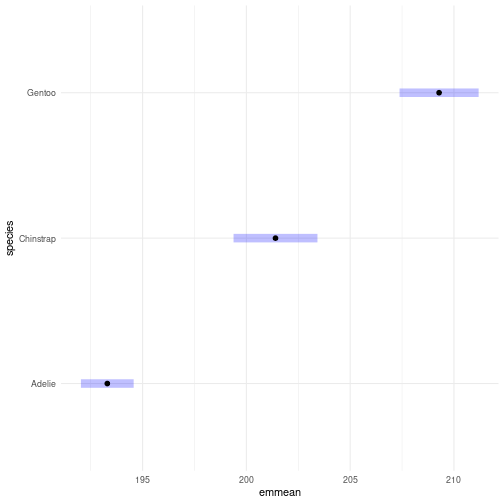<!-- -->