TD2 : Charger, manipuler et visualiser des données dans R
UP Mathématiques appliquées
Objectifs de la séance
- Manipulation de données
- Sélectionner une partie du jeu de données :
- sélectionner des variables (colonnes),
- sélectionner des individus (lignes),
- utiliser un critère de sélection.
- Sélectionner une partie du jeu de données :
- Visualisation de données
- Représentation du lien entre une variable quantitative et une variable qualitative
- Représentation du lien entre deux variables quantitatives
- contrôle de la transparence et des couleurs
- personnalisation des graphiques
- Analyse de données
Exercices
Retour sur la séance de TD 1 : de l’intérêt de créer un projet
Au cours de la dernière séance, vous avez créé un projet dans Rstudio et enregistré un script R qui permet notamment d’importer des données. Le TD d’aujourd’hui s’inscrit dans la continuité du précédent et propose de poursuivre la découverte du jeu de données des manchots de Palmer.
Aller dans le répertoire ds_TD et cliquer sur l’icone associé au projet créé lors de la dernière séance. Rstudio s’ouvre et reprend apparemment la session là où elle s’était arrêtée :
- la fenêtre Environment, en haut à droite, indique que l’objet penguins a été sauvegardé,
- la fenêtre Files, en bas à droite, indique que le script R
TD1_script.Ra été sauvegardé.
Ouvrir le script de la dernière séance et l’enregistrer sous le nom TD2_script.R. La fenêtre Files liste maintenant deux fichiers R dans votre répertoire ds_TD, un pour chacune des séances de TD, le script de la deuxième séance incluant les lignes de commande de celui de la première.
Cliquer sur la fenêtre Packages.
- Que constatez-vous ?
Rstudio n’a chargé aucun package à l’ouverture du projet et il faut les charger via la fonction library (vue en TD1) dont l’argument principal est le nom du package souhaité.
- Exécuter à nouveau la ligne qui charge le package
tidyverse. Dans l’onglet Packages, la ligne portant sur tidyverse est maintenant cochée.
Le tidyverse est en fait un ensemble de packages, contenant entre autres
- le package
dplyrdédié à la manipulation de données, - le package
ggplot2dédié à la visualisation de données.
Manipulation de données dans R
Sélectionner des variables (opérations sur des colonnes)
La fonction select du package dplyr permet de sélectionner des variables (des colonnes) d’un jeu de données.
Quels arguments faut-il renseigner pour sélectionner la seule variable
Espece?Sélectionner cette variable puis l’afficher.
Sélectionner cette variable et la stocker dans un objet nommé
dta.Que fait la commande suivante ?
select(penguins, -poids)Cette opération peut se comprendre de la façon suivante : à partir du jeu de données penguins, on retire la variable poids. Ceci peut s’écrire à l’aide d’un opérateur appelé pipe et noté %>% (raccourci CTRL + Shift + m ), de la façon suivante :
penguins %>% select(-poids)L’opérateur pipe permet d’enchaîner les commandes et préserve la lisibilité.
- Que fait la suite de commandes suivante ?
penguins %>%
select(-poids) %>%
select(-ile) %>%
select(-espece)- Créer un nouveau jeu de données à partir du jeu de données penguins, en supprimant la variable
poids.
Il est possible de sauvegarder ce jeu de données simplifié dans l’objet penguins lui-même, c’est ce qu’illustre la ligne de commande suivante :
penguins <- penguins %>% select( -poids)Sélectionner des individus (opérations sur les lignes)
- Consulter l’aide de la fonction
filter - Sélectionner les individus pour lesquels la longueur de bec dépasse 35mm
De façon formelle, vous venez d’utiliser l’opérateur logique >. Les opérateurs logiques permettent notamment d’exprimer des conditions. Les plus importants sont, in english :
<less than<=less than or equal to>greater than>=greater than or equal to==exactly equal to!=not equal to!xNot xx | yx OR yx & yx AND y
Remarque : pour transformer vos variables, vous utiliserez des opérateurs arithmétiques tels que +, pour additionner deux variables par exemple.
- Dans le jeu de données penguins, le sexe de certains individus n’est pas renseigné (on parle de données manquantes - Not Available en anglais). On peut identifier ces individus grâce à la commande
penguins %>% filter(is.na(sexe))- A l’aide de l’opérateur
!x, garder uniquement les individus pour lesquels le sexe est renseigné
Combiner les sélections d’individus et de variables
- Combiner plusieurs conditions : à partir du jeu de données penguins, garder les individus pour lesquels le sexe est renseigné, conserver les variables longueur du bec, longueur des nageoires et sexe, stocker votre sélection dans un objet que vous appellerez manchots.
Visualisation de données
Grâce au jeu de données manchots, il est possible de comparer les longueurs de bec chez les mâles et les femelles.
Comparer des distributions à l’aide d’histogramme
- Proposer un graphique pour visualiser la distribution des longueurs de bec, sans prendre en compte le sexe
Pour différencier les distributions des longueurs de bec selon le sexe, il est nécessaire d’ajouter une information sur le sexe sur le graphique créé précédemment, comme proposé dans la commande suivante :
manchots %>% ggplot() + geom_histogram(aes(x=longueur_bec, fill = sexe))- Discuter du choix par défaut pour représenter la variable sexe
Pour obtenir un histogramme par sexe, il faut modifier le comportement par défaut et indiquer que l’on veut deux histogrammes différents et non pas superposer dans chaque classe le nombre de mâles et de femelles. Il faut modifier l’argument position par défaut.
manchots %>% ggplot() + geom_histogram(aes(x=longueur_bec, fill = sexe), position = 'identity')Enfin pour bien visualiser les différents histogrammes, il est possible de jouer sur la transparence.
manchots %>% ggplot() + geom_histogram(aes(x=longueur_bec, fill = sexe), position = 'identity', alpha = 0.6)- (En option) Refaire ce même graphique en représentant les densités par classes.
Comparer des distributions à l’aide de boîtes à moustaches
Il est difficile, à l’oeil, de comparer des histogrammes : les boîtes à moustaches sont plus adaptées pour comparer l’effet du sexe sur la distribution des longueurs de bec.
- Rappeler la nature des variables
sexeetlongueur_bec.
Les boîtes à moustaches sont une représentation adaptée à la visualisation de l’effet d’une variable qualitative sur une variable quantitative, elles sont obtenues dans R grâce à la fonction graphique geom_boxplot. Il faut spécifier que l’abscisse est la variable qualitative et l’ordonnée la variable quantitative.
- Représenter à l’aide de boîtes à moustaches les distributions des longueurs de bec en fonction du sexe.
Ce graphique par défaut est largement personnalisable (cf fin du TD)
Visualiser le lien entre taille du bec et longueur des nageoires
Rappeler la nature des variables longueur du bec et longueur des nageoires. Quelle sorte de graphique semble le plus adpatée ?
Utiliser la fonction graphique
geom_pointen précisant bien les abscisses et les ordonnées pour obtenir une visulaisation du lien entre la longueur des nageoires sur la longueur du bec.Utiliser la couleur pour différencier les mâles des femelles.
Ajouter un titre au graphique précédent
Personnalisation des graphiques
Il est possible de modifier l’intitulé par défaut des axes grâce à la fonction labs.
manchots %>% ggplot() +
geom_boxplot(aes(x = sexe, y = longueur_bec)) +
labs(x ='Sexe', y = 'Longueur du bec (mm)' )Il est également possible de représenter les données elles-mêmes. On peut utiliser la commande geom_point qui va dessiner un point aux abscisses et ordonnées spécifiées.
manchots %>% ggplot() +
geom_boxplot(aes(x = sexe, y = longueur_bec)) +
labs(x ='Sexe', y = 'Longueur du bec (mm)' ) +
geom_point(aes(x = sexe, y = longueur_bec)) Le résultat n’est pas très satisfaisant, car tous les points ayant la même abscisse, il est difficile de les différencier. Il est possible de perturber légèrement l’abscisse de manière à mieux distinguer les différentes mesures.
manchots %>% ggplot() +
geom_boxplot(aes(x = sexe, y = longueur_bec)) +
labs(x ='Sexe', y = 'Longueur du bec (mm)' ) +
geom_jitter(aes(x = sexe, y = longueur_bec)) Finalement pour avoir un très joli graphique, il est possible de
- limiter la perturbation de l’abscisse,
- dessiner des points plus petits,
- les rendre légèrement transparents.
manchots %>% ggplot() +
geom_boxplot(aes(x = sexe, y = longueur_bec)) +
labs(x ='Sexe', y = 'Longueur du bec (mm)' ) +
geom_jitter(aes(x = sexe, y = longueur_bec), width = 0.3, size=0.4, alpha=0.8) Utiliser la couleur comme cerise sur le gâteau.
manchots %>% ggplot() +
geom_boxplot(aes(x = sexe, y = longueur_bec, fill = sexe), alpha = 0.2) +
labs(x ='Sexe', y = 'Longueur du bec (mm)' ) +
geom_jitter(aes(x = sexe, y = longueur_bec, col = sexe), width = 0.3, size=0.4, alpha=0.8) +
labs(fill = 'Sexe', col = 'Sexe')Reprendre les graphiques qui visualisent l’effet de la longueur des nageoires sur la longueur du bec.
- La relation liant les deux variables s’apparente-t-elle à une droite ?
manchots %>% ggplot() +
geom_point(aes(x = longueur_nageoire, y = longueur_bec)) +
xlab('Longueur des nageoires (mm)') +
ylab('Longueur du bec (mm)') +
geom_smooth(method = 'lm', aes(x = longueur_nageoire ,
y = longueur_bec))- Cette relation est-elle la même pour les mâles et les femelles ?
manchots %>% ggplot() +
geom_point(aes(x = longueur_nageoire, y = longueur_bec, col = sexe)) +
xlab('Longueur des nageoires (mm)') +
ylab('Longueur du bec (mm)') +
geom_smooth(method = 'lm', aes(x = longueur_nageoire ,
y = longueur_bec, col = sexe))La fonction aes étant utilisé 2 fois de façon équivalente, il est possible de factoriser la fonction aes
manchots %>% ggplot() +
aes(x = longueur_nageoire, y = longueur_bec, col = sexe) +
geom_point() +
xlab('Longueur des nageoires (mm)') +
ylab('Longueur du bec (mm)') +
geom_smooth(method = 'lm')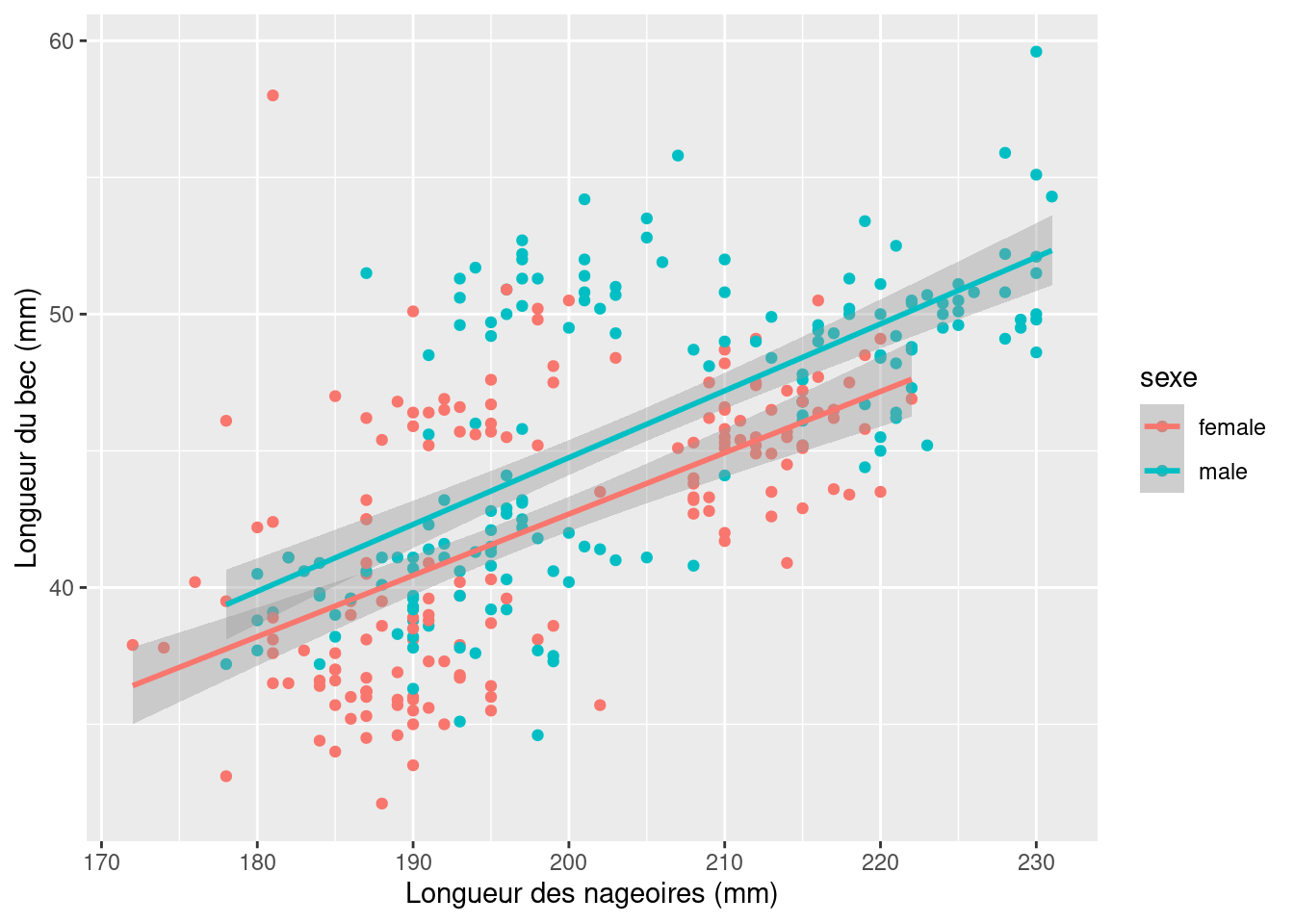
Terminer une session de travail
Il est essentiel de nettoyer (ne garder que les commandes pertinentes) et de sauvegarder le script final pour pouvoir le réutiliser lors d’une séance prochaine. Il est préférable de charger les packages lors des premières lignes du script.
- Enregistrer votre script avant de quitter
R.
Le vocabulaire de la séance
Commandes R
- %>%
- filter
- geom_boxplot
- geom_jitter
- geom_point
- geom_smooth
- is.na
- labs
- select
Environnement R
- Reprendre un projet
Statistique
- distribution d’une variable